在数据分析中,K-means 聚类是一种非常常用的聚类方法。它的核心思想是:将相似的数据点归为同一簇,并为每个簇计算一个质心,然后把每个点分配到距离最近的质心所属簇中。
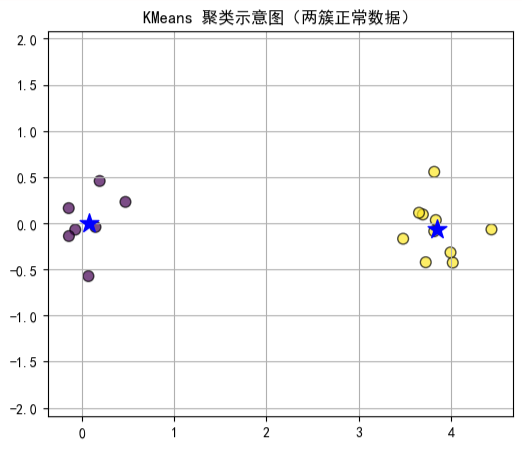
这种方法简单高效,但也存在一些明显的局限性:
- 对噪声和异常点敏感:极端值会影响质心的位置,从而扭曲聚类结果。
- 需要预先指定簇的数量:在实际场景中,簇的数量往往未知,而错误的预设可能导致聚类不准确。
- 对复杂数据分布适应性差:对于非球形或不规则形状的簇,K-means 很难得到理想的划分。
如下图所示,我们可以看到 K-means 在某些复杂数据分布下的聚类效果并不理想。
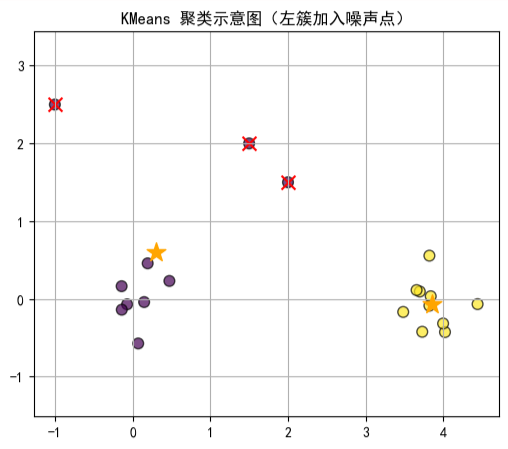
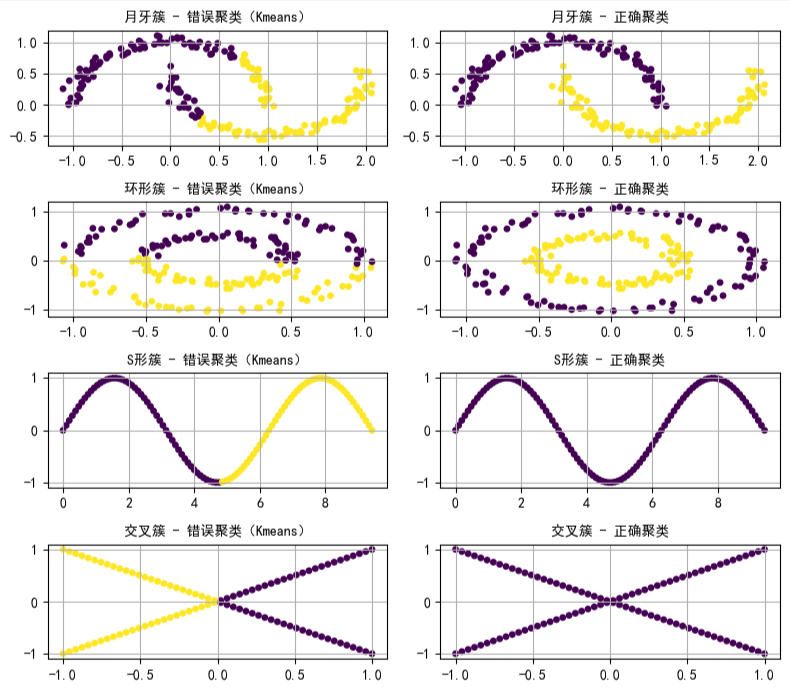
为了克服这些问题,DBSCAN(Density-Based Spatial Clustering of Applications with Noise)应运而生。它是一种基于密度的聚类方法,相比 K-means 有几个明显优势:
- 无需预先指定簇的数量,能够自动发现簇的数量。
- 能够识别任意形状的簇,不局限于球形。
- 自动识别噪声点,避免异常点干扰聚类结果。
这里的密度指的是单位区域内点的多少,简单来说,DBSCAN 就是找点多的地方,把它们聚成簇,把点少的地方当作噪声。
凭借这些特点,DBSCAN 在地理信息分析、图像处理、数据挖掘等领域得到了广泛应用。
其他链接:
基于质心的聚类方法:https://mengbaoliang.cn/archives/93699/
基于分布的聚类方法:https://mengbaoliang.cn/archives/tag/gshh/
1. 算法原理
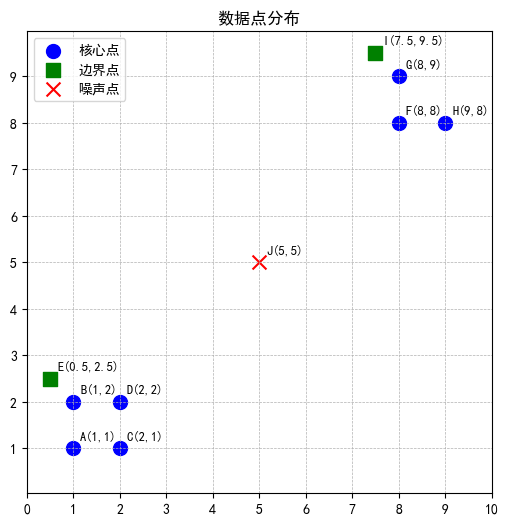
在理解 DBSCAN 聚类原理之前,先要明确一点,在聚类的过程中,DBSCAN 会把所有数据点分成三类:
- 核心点:在半径 epsilon 范围内至少有 min_samples 个点(算上自己)
- 边界点:自己周围点数不够 min_samples,但落在某个核心点的 epsilon 范围内
- 噪声点:既不是核心点,也不在任何核心点的 epsilon 范围内
| A | B | C | D | E | F | G | H | I | J |
|---|---|---|---|---|---|---|---|---|---|
| (1,1) | (1,2) | (2,1) | (2,2) | (0.5,2.5) | (8,8) | (8,9) | (9,8) | (7.5,9.5) | (5,5) |
假设:epsilon = 1.5、min_samples = 3
接下来,我们看看具体的聚类过程。计算每个数据点到其他数据点的距离:
| A | B | C | D | E | F | G | H | I | J | |
|---|---|---|---|---|---|---|---|---|---|---|
| A | 0.000 | 1.000 | 1.000 | 1.414 | 1.581 | 9.899 | 10.630 | 10.630 | 10.700 | 5.657 |
| B | 1.000 | 0.000 | 1.414 | 1.000 | 0.707 | 9.220 | 9.899 | 10.000 | 9.925 | 5.000 |
| C | 1.000 | 1.414 | 0.000 | 1.000 | 2.121 | 9.220 | 10.000 | 9.899 | 10.124 | 5.000 |
| D | 1.414 | 1.000 | 1.000 | 0.000 | 1.581 | 8.485 | 9.220 | 9.220 | 9.301 | 4.243 |
| E | 1.581 | 0.707 | 2.121 | 1.581 | 0.000 | 9.301 | 9.925 | 10.124 | 9.899 | 5.148 |
| F | 9.899 | 9.220 | 9.220 | 8.485 | 9.301 | 0.000 | 1.000 | 1.000 | 1.581 | 4.243 |
| G | 10.630 | 9.899 | 10.000 | 9.220 | 9.925 | 1.000 | 0.000 | 1.414 | 0.707 | 5.000 |
| H | 10.630 | 10.000 | 9.899 | 9.220 | 10.124 | 1.000 | 1.414 | 0.000 | 2.121 | 5.000 |
| I | 10.700 | 9.925 | 10.124 | 9.301 | 9.899 | 1.581 | 0.707 | 2.121 | 0.000 | 5.148 |
| J | 5.657 | 5.000 | 5.000 | 4.243 | 5.148 | 4.243 | 5.000 | 5.000 | 5.148 | 0.000 |
统计每个数据点的邻域
| 数据点 | 邻域点集合 | 邻域点数量 | 数据点类型 |
|---|---|---|---|
| A(1,1) | A, B, C, D | 4 | 核心点 |
| B(1,2) | A, B, C, D, E | 5 | 核心点 |
| C(2,1) | A, B, C, D | 4 | 核心点 |
| D(2,2) | A, B, C, D | 4 | 核心点 |
| E(0.5,2.5) | B, E | 2 | 边界点 |
| F(8,8) | F, G, H | 3 | 核心点 |
| G(8,9) | F, G, H, I | 4 | 核心点 |
| H(9,8) | F, G, H | 3 | 核心点 |
| I(7.5,9.5) | G, I | 2 | 边界点 |
| J(5,5) | J | 1 | 噪声点 |
开始聚类:
第一步:外层循环遍历数据点
- 访问 A:A 未访问且为核心点 → 创建簇 C1
- 将 A 的邻域 {A, B, C, D} 全部加入簇
- 核心邻居 B、C、D 入队列等待扩展 → 队列状态 [B, C, D]
第二步:内层队列扩展簇 C1
- 出队 B:邻域 {B, E, A, D, C}
- E 不是核心点,但在 B 的邻域内 → 加入簇作为边界点,不入队
- 队列更新为 [C, D]
- 出队 C:邻域 {C, A, D, B} 已全部在簇中 → 无新增,队列 [D]
- 出队 D:邻域 {D, B, C, A} 已全部在簇中 → 队列清空,C1 扩展完成
第三步:外层循环继续遍历
- 遇到 B、C、D、E 已访问 → 跳过
- 下一个未访问核心点 F → 创建簇 C2
- 将 F 的邻域 {F, G, H} 加入簇
- 核心邻居 G、H 入队列 → 队列状态 [G, H]
第四步:内层队列扩展簇 C2
- 出队 G:邻域 {G, I, F, H}
- I 不是核心点,但在 G 的邻域内 → 加入簇作为边界点,不入队
- 队列更新为 [H]
- 出队 H:邻域 {H, F, G} 已全部在簇中 → 队列清空,C2 扩展完成
第五步:外层循环继续遍历剩余数据点
- I 已访问 → 跳过
- J 未访问且邻域点数 < min_samples,且不在任何核心点邻域 → 判定为噪声点
第六步:最终聚类结果
- 簇 C1:核心点 A、B、C、D;边界点 E
- 簇 C2:核心点 F、G、H;边界点 I
- 噪声点:J
通过对 DBSCAN 聚类过程的了解,我们可以发现,簇的形成和扩展是依靠核心点推动的,而边界点会被吸收进簇,噪声点则不属于任何簇。
简单总结下,在 DBSCAN 里,簇是长出来的,而不是一开始就分好的。整个聚类的过程可以想成:
- 找到一个核心人物(邻居够多的点)。
- 这个核心人物会把自己周围(epsilon 范围内)的邻居都拉进来。
- 新进来的点如果自己也是“核心”,就继续往外扩 → 簇就这样不断生长。
- 如果一个点既不是核心,也没有核心愿意拉它,就成了噪声。
另外,我们也发现 DBSCAN 聚类过程中,寻找邻域需要大量的计算。例如:要对 1000 条数据进行聚类,则会进行 100 万次的距离计算,如果数据维度较高,则会需要更多的计算时间。所以,DBSCAN 的聚类优化,主要是通过对邻域计算的优化来提升聚类效率。常见的是,使用 KD TREE 或者 BALL TREE,KD Tree 适合低维数据(通常 20 维),BALL tree 适合较高维数据(20 维以上)。
2. 样本归簇
在很多聚类算法中,例如 K-Means 和 高斯混合模型(GMM),训练结束后通常会提供 predict 接口。当新样本到来时,无需依赖原始数据的完整分布,只需基于模型训练得到的簇的数学描述,即可直接判定新样本的簇归属。例如:
- K-Means:训练后会得到各簇的质心,新样本的归属可通过计算其与各质心的距离确定
- 高斯混合模型(GMM):训练后会输出每个高斯子分布的均值、方差、混合系数,新样本的归属可通过计算其在各子分布下的概率密度,选择概率最大的簇作为结果
但 DBSCAN 与二者存在本质区别:DBSCAN 的核心逻辑是基于密度划分簇,它仅对已有训练数据点完成标记,例如:哪些点属于某一簇、哪些点是噪声点,并不会生成质心或者概率分布这类可描述簇形态的数学参数。
因此,DBSCAN 本质上是一种非参数化、完全依赖原始数据密度分布的聚类算法:由于缺乏对簇的通用数学描述,它无法像 K-Means 或 GMM 那样,直接通过 predict 接口判定新样本的簇归属(若要处理新样本,需将其重新放入原始数据集,结合密度规则重新计算)。
3. 设置参数
DBSCAN 聚类需要两个非常关键的超参数:epsilon(邻域半径) 和 min_samples(核心点的最少邻居数)。
这两个参数的选择会直接影响聚类效果。虽然没有一个固定的“最佳公式”,但有一些直观的经验方法可以帮助我们更合理地设置。
在实践中,通常建议 先确定 min_samples,再选择合适的 epsilon。
3.1 min_samples
min_samples 最常用的参考公式为:
min_samples ≈ 数据维度 + 1
这样设置的直观意义是:在 d 维空间中,核心点的邻域至少需要 d+1 个点,才能形成一个稳定的几何结构,从而保证簇的密度足够高。举几个例子:
- 1 维空间:1 维空间是直线,要形成密集段,一个点至少需要 1 个相邻点(加上自身共 2 个)
- 2 维空间:2 维空间是平面,要形成密集区域,至少需要 3 个点才能构成最小三角形单元
- d 维空间:需要 d+1 个点才能支撑起 d 维空间的最小密集结构,避免因点数量过少导致误判密集区域
注意:这个公式并不是硬性规定,只是一种基于几何直觉的经验参考。实际中,还需要结合数据分布进行调整。
注意:这只是一个 “入门级参考标准”,并非绝对准则。实际应用中,若数据噪声较多,可适当增大 min_samples(如取 d+2、d+3),减少噪声点被误判为核心点的概率;若数据簇本身较稀疏,可适当减小 min_samples,避免漏判核心点。
3.2 epsilon
当 min_samples 确定后,epsilon 的作用就是划定邻域范围,确保核心点的邻域内,能稳定包含至少 min_samples 个样本。此时最有效的方法是K 距离图法,其中 “K 值” 需与已确定的 min_samples 保持一致,即 K=min_samples。
下面举个例子,假设有 5 个数据点,k = 3(min_samples = 3)
A(1,1), B(2,2), C(2,0), D(5,5), E(8,8)
计算每个点到其他点的距离:
| 点 | A | B | C | D | E |
|---|---|---|---|---|---|
| A | 0 | 1.41 | 1.41 | 5.66 | 9.90 |
| B | 1.41 | 0 | 2.00 | 4.24 | 8.49 |
| C | 1.41 | 2.00 | 0 | 5.83 | 10.00 |
| D | 5.66 | 4.24 | 5.83 | 0 | 4.24 |
| E | 9.90 | 8.49 | 10.00 | 4.24 | 0 |
对每个点,把距离排序,取第 3 个最近邻距离(k = 3)
| 点 | 升序距离(不含自己) | 第 3 近邻距离 |
|---|---|---|
| A | 1.41, 1.41, 5.66, 9.90 | 5.66 |
| B | 1.41, 2.00, 4.24, 8.49 | 4.24 |
| C | 1.41, 2.00, 5.83, 10.00 | 5.83 |
| D | 4.24, 4.24, 5.66, 5.83 | 5.66 |
| E | 4.24, 8.49, 9.90, 10.00 | 9.90 |
排序第 3 近邻距离:4.24、5.66、5.66、5.83、9.90,并绘制 K 距离图:
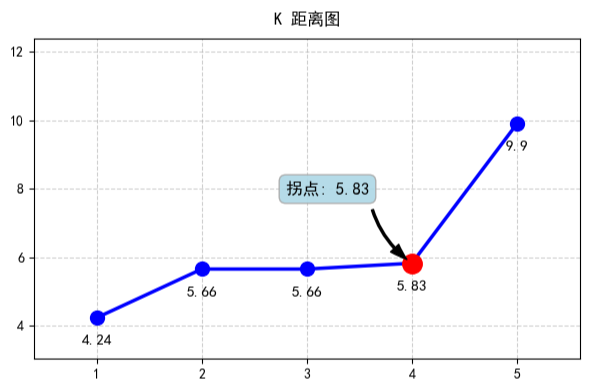
在上图中:
- K 距离较小的点,说明它周围邻居密集
- K 距离较大的点,说明点周围邻居稀疏
- 密集区域和稀疏区域之间的点称作拐点
拐点左侧是高密度区域,右侧是低密度区域,因此 epsilon 应取拐点附近的区间,避免将低密度样本误判为核心点。上图这种情况,我们可以大概在 [5.66, 5.83] 之间选择一个作为 epsilon 的值,这样能让 DBSCAN 准确识别核心点和噪声点,得到合理的聚类结果。
注意:即使初步选择的 epsilon 值参考了 K 距离图,聚类效果可能仍存在不足,此时可以通过微调 epsilon 和 min_samples,以获得更合理的簇划分。
4. 接口使用
import numpy as np
from sklearn.cluster import DBSCAN
import pandas as pd
def demo():
points = np.array([[1, 1], [1, 2], [2, 1], [2, 2], [0.5, 2.5], [8, 8], [8, 9], [9, 8], [7.5, 9.5], [5, 5]])
columns = ['A', 'B', 'C', 'D', 'E', 'F', 'G', 'H', 'I', 'J']
# algorithm 邻域搜索算法,可选值:auto、ball_tree、kd_tree、brute
# metric 样本距离计算方法,例如:euclidean、cosine、minkowski、manhattan
dbscan = DBSCAN(eps=1.5, min_samples=3, metric='euclidean', algorithm='brute')
result = dbscan.fit_predict(points)
# 聚类结果
# [ 0 0 0 0 0 1 1 1 1 -1]
# 0 表示第一个簇
# 1 表示第二个簇
# -1 表示噪声点
print(result)
# 核心样本索引:[0 1 2 3 5 6 7]
print(dbscan.core_sample_indices_)
# 具体核心样本:[[1. 1.] [1. 2.] [2. 1.] [2. 2.] [8. 8.] [8. 9.] [9. 8.]]
print(dbscan.components_)
if __name__ == '__main__':
demo()




 冀公网安备13050302001966号
冀公网安备13050302001966号